IA mapeia 200 milhões de proteínas e resolve desafio de 50 anos na ciência
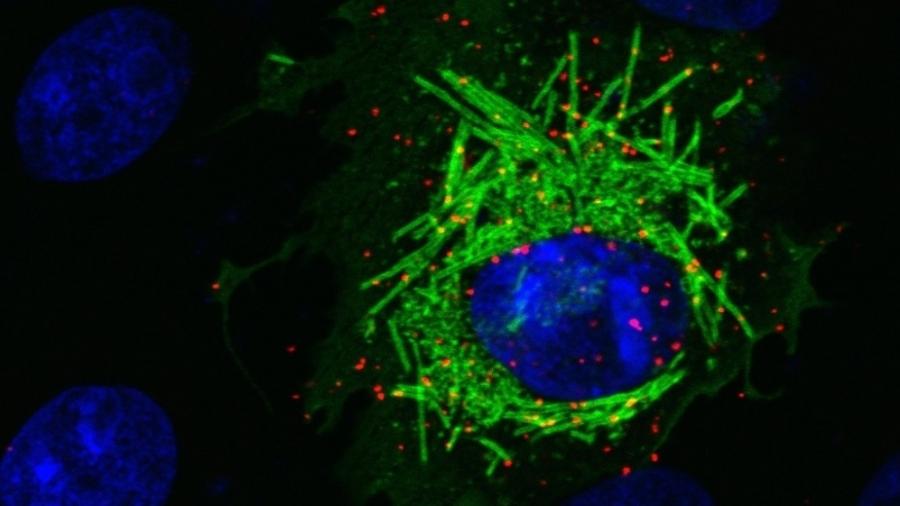
AlphaFold, a Inteligência Artificial criada pela empresa DeepMind, conseguiu analisar e catalogar mais de 200 milhões de proteínas - praticamente todas as que existem, segundo cientistas.
Nos últimos 50 anos, identificar essas estruturas havia sido um dos maiores desafios da biologia. A IA conseguiu fazer o trabalho em apenas 18 meses, um tempo considerado recorde para a ciência.
O número foi revelado pela DeepMind nesta semana, ao anunciar a ampliação do acesso gratuito ao AlphaFold Protein Structure Database (Banco de Dados de Estruturas de Proteína da AlphaFold) para pesquisadores do mundo todo.
Segundo a empresa, os modelos elaborados pela IA já foram citados em mais de 4 mil estudos científicos desde que ela foi criada, em 2020.
O trabalho desses dois anos foi realizado em parceria com o Instituto Europeu de Bioinformática do Laboratório Europeu de Biologia Molecular (EMBL-EBI). As informações sobre as estruturas das proteínas também foram publicadas no UniProt, repositório de pesquisa de proteínas mundialmente reconhecido.
A DeepMind é o braço de inteligência artificial da Alphabet, empresa controladora do Google, com sede no Reino Unido.
'Presente para a humanidade'
Um dos cientistas que trabalhou no projeto, Ewan Birney, da EMBL-EBI, chegou a dizer que considera o AlphaFold Database "um presente para a humanidade".
"Como alguém que trabalha com genômica e biologia computacional desde a década de 1990, vi muitos desses momentos em que você pode sentir a paisagem mudando sob você e o fornecimento de novos recursos, e este foi um dos mais rápidos. Há dois anos, simplesmente não percebemos que isso era viável", disse Birney, em entrevista à publicação Newscientist.
Com esse avanço, a AlphaFold revolucionou os conhecimentos em biologia e ciência básica, o que vai permitir que os cientistas entendam melhor a evolução de muitas doenças e desenvolvam novos medicamentos e novos produtos.
Segundo a Newscientist, o banco de dados permitiu avanços em algumas áreas, como no combate à malária, bactérias resistentes a antibióticos e enzimas para quebrar resíduos plásticos.
Proteínas são estruturas de difícil compreensão
As proteínas são feitas de longas fitas de aminoácidos. Alguns deles são atraídos por outros, outros repelidos pela água e as cadeias vão se contorcendo e criando formas complexas que são difíceis de determinar com precisão. Acessar esse enovelamento é fundamental para compreender muitas doenças ou mesmo para combater algumas espécies de pragas.
No caso da malária, por exemplo, entendendo a proteína do inseto é possível quebrar o ciclo de vida deles. Por isso, tanto a IA como o banco de dados foram passos tão importantes para a ciência.
Em entrevista ao MIT Technology Review, Hassabis afirmou que o enovelamento de proteínas é um problema que ele tentava resolver há mais de 20 anos.
"Eu diria que esta é a maior coisa que fizemos até agora. É o mais empolgante de certa forma, porque deve ter o maior impacto no mundo fora da inteligência artificial", completou.
Quais os próximos passos?
Os cientistas reconhecem a importância do AlphaFold mas admitem que agora é hora de avançar em novos passos e resolver outras questões relacionadas às proteínas.
Para alguns cientistas, a IA tem suas limitações e é preciso melhorar a precisão da ferramenta, desenvolvendo um modelo que compreenda como as proteínas se dobram e não apenas sua estrutura final.
Em entrevista à Newscientist, Keith Willison, do Imperial College London, afirmou que o AlphaFold não é capaz de pegar qualquer sequência arbitrária de aminoácidos e modelar exatamente na forma como se dobram. Existem também as interações complexas e desordenadas entre as proteínas, que não têm padrões, e que também não foram resolvidas ainda pela IA.
A ferramenta só é capaz de usar as partes de proteínas e suas estruturas que foram determinadas experimentalmente para prever como uma nova proteína se dobrará. Ou seja, suas estruturas são sempre previsões e não resultados calculados.
Pushmeet Kohli, que lidera a equipe científica da DeepMind, afirma que a empresa está trabalhando para melhorar a precisão e os recursos. "Conhecemos a estrutura estática das proteínas, mas não é aí que o jogo termina", reconheceu.
"Queremos entender como essas proteínas se comportam, qual é sua dinâmica, como elas interagem com outras proteínas. Depois, há a outra área da genômica onde queremos entender como a receita da vida se traduz em quais proteínas são criadas, quando são criadas e o funcionamento de uma célula", detalhou.
















ID: {{comments.info.id}}
URL: {{comments.info.url}}
Ocorreu um erro ao carregar os comentários.
Por favor, tente novamente mais tarde.
{{comments.total}} Comentário
{{comments.total}} Comentários
Seja o primeiro a comentar
Essa discussão está encerrada
Não é possivel enviar novos comentários.
Essa área é exclusiva para você, assinante, ler e comentar.
Só assinantes do UOL podem comentar
Ainda não é assinante? Assine já.
Se você já é assinante do UOL, faça seu login.
O autor da mensagem, e não o UOL, é o responsável pelo comentário. Reserve um tempo para ler as Regras de Uso para comentários.